Actualidad informática
Noticias y novedades sobre informática
Oreja artificial mediante impresión 3D
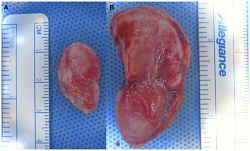
La Universidad Cornell (EE.UU.) ha dado a conocer un trabajo de investigación en el que han desarrollado una oreja artificial apoyándose en la impresión 3D y en el uso de células de cartílago natural. Gracias a este avance, los científicos esperan poder realizar el primer implante de estas características en el año 2016.
Para empezar, el equipo de investigadores trabajaron en un CAD con el que modelaron una representación tridimensional de la oreja del paciente que tomaba como base los datos captados al realizar escáneres en 3D de orejas de niños que no presentaban microtia (aunque la idea es que, en el futuro, se pueda generar el modelo de la oreja escaneando la oreja sana al paciente y generando una imagen especular de la misma).
A partir de ahí, el siguiente paso fue crear un molde mediante impresión 3D y, acto seguido, lo rellenaron con un gel de colágeno en el que introdujeron células de cartílago (células condrógenas) procedentes de una vaca pero la idea es que, en una hipotética aplicación real, la fuente de las células sería el mismo paciente. Alimentando el molde con nutrientes, las células codrógenas se reproducen y van reemplazando, paulatinamente, al colágeno hasta formar una estructura más o menos estable que se puede suturar al paciente y recubrir con piel mientas se sigue desarrollando y se sustituye por completo el colágeno para dejarle espacio a las células condrógenas. En el caso del experimento realizado por los investigadores, la oreja se suturó al lomo de una rata de laboratorio donde se terminó de desarrollar tras tres meses de proceso.
Aunque aún quedan algunos aspectos a depurar en esta investigación, el equipo ve viable realizar un implante real en el año 2016, fecha en la que esperan certificar la técnica con humanos (tras realizar ensayos clínicos).
Ampliar en: ALT1040
Bajo licencia Craetive Commons
Cuatro gramos de ADN bastarían para almacenar la información del planeta

Investigadores han logrado crear un proceso con el que sería posible almacenar sin errores datos en la forma de cadenas sintéticas de ADN, las cuales en teoría podrían perdurar intactas por miles de años. De acuerdo con sus cálculos, el método de codificación de información desarrollado marcaría la posibilidad de guardar cerca de cien millones de horas de video en Alta Definición, o incluso contener toda la información del mundo, equivalente a 1.8 zettabytes, en tan sólo cuatro gramos de peso.
Hace unos días se mostraban los grandes avances que marcaban este proceso como una realidad prácticamente inevitable y ahora aquí presentamos la primera prueba. El estudio, publicado en la revista de investigación Nature esta misma semana, fue desarrollado por un grupo de científicos británicos del European Bioinformatics Institute EMBL-EBI, quienes indican haber podido almacenar una versión MP3 del famoso discurso de Martin Luther King “I Have a Dream” junto a una imagen del logotipo de la institución y algunos archivos de texto (probablemente listas del súper), dentro de una cadena sencilla de ADN.
Fuente: AlT1040
Almacenamiento de prosa y recuperación libre de errores en ADN
Es uno de los discursos más emblemáticos de todos los tiempos, y ahora ha sido inmortalizado de una forma muy inusual. Un fragmento de 1963 de Martin Luther King «I have a dream» (Tengo un sueño) ha sido almacenado en el alfabeto del ADN.
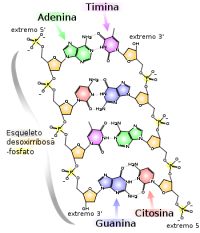
Nick Goldman en el European Bioinformatics Institute en Hinxton, Reino Unido, y sus colegas sintetizaron ADN para codificar una mezcla ecléctica de información en sus componentes adenina, timina, citosina y guanina. Utilizaron estas «letras» para grabar un archivo de audio de 26 segundos del discurso de King, los 154 sonetos de Shakespeare, una foto digital de su laboratorio y el famoso artículo en el que James Watson y Francis Crick describieron por primera vez la estructura de doble hélice del ADN .
El equipo construyó anteriormente técnicas de codificación de ADN añadiendo corrección de errores, permitiendo la recuperación con el 100 por cien de precisión.
Memoria basada en el ADN es eficaz dado que el ADN puede durar miles de años sin condiciones especiales de almacenamiento, aparte de encontrarse en algún lugar frío, oscuro y seco. En teoría, el ADN puede codificar o menos la capacidad de 100 000 millones de DVD por gramo de ADN de cadena sencilla, por lo que es potencialmente útil para almacenar la enorme cantidad de datos archivados producidos por lugares como el CERN.
La falsa creencia de que solo usamos el 10% de nuestro cerebro
Todos habrán oído que sólo usamos el 10% del cerebro. La evidencia dice lo contrario, tal como explica Barry Beyerstein en el libro Mind Myths: Exploring Popular Assumptions About the Mind and Brain de Sergio Della Sala.
Así está resumido en Wikipedia:
- Estudios sobre el daño cerebral: Si el 90% del cerebro no se utiliza, entonces cuando se lesionan ciertas áreas no debe afectar al rendimiento. En cambio, no hay ningún área del cerebro que pueda ser dañada sin que se pierda alguna habilidad. Incluso los daños en las áreas más pequeñas pueden conllevar consecuencias graves.
- Evolución: El cerebro necesita un enorme gasto energético en comparación con el resto del cuerpo, consume una gran cantidad de oxígeno y nutrientes. Si el 90% del mismo no fuese necesario los humanos con el cerebro más pequeño tendrían grandes ventajas para sobrevivir, ya que sus cerebros serían más eficientes. Así que el proceso de selección natural debería haber eliminado los cerebros ineficientes.
- Imágenes cerebrales: Tecnologías como la tomografía por emisión de positrones (PET) y la imagen por resonancia magnética nuclear funcional (fMRI) permiten monitorizar la actividad cerebral de personas vivas. Estas técnicas han revelado que, incluso mientras dormimos, todas las partes del cerebro presentan algún nivel de actividad. Sólo cuando el cerebro sufre un daño grave tiene “silenciadas” algunas áreas.
- Localización de función: En lugar de trabajar como una sola masa, el cerebro tiene regiones distintas para los diferentes tipos de procesamiento de la información. Varias décadas de investigación han permitido mapear las funciones de las áreas del cerebro, y no se han encontrado áreas que no tengan ninguna función.
- Análisis microestructural: Mediante la técnica de grabación de unidades individuales (single-unit recording), los investigadores han insertado un electrodo diminuto en el cerebro para monitorizar la actividad de una sola célula. Si no se utilizan el 90% de las células, esta técnica lo debería haber demostrado.
- Estudios metabólicos: Otra técnica científica implica estudiar la adopción de moléculas de 2-desoxi-D-glucosa etiquetadas radiactivamente en el cerebro. Si el 90 % del cerebro estuviera inactivo, entonces esas células inactivas deberían aparecer como áreas en blanco en una radiografía del cerebro. Una vez más, no hay tal resultado.
- Enfermedades neuronales: Las células del cerebro que no se utilizan deberían degenerarse. Por lo tanto, si el 90% del cerebro permaneciera inactivo, las autopsias de cerebros adultos tendrían que revelar una degeneración a gran escala.
Fuente: DE AVANZADA
Impresora 3D capaz de “imprimir” cartílago

Un grupo de investigadores estadounidenses ha desarrollado un híbrido de impresora capaz de imprimir cartílago. Un dispositivo que mezcla la tecnología de inyección de tinta modificada con una máquina de electrospinning, combinando células cartilaginosas con estructuras artificiales. Una impresora que en el futuro podrá ser utilizada como implante en pacientes con lesiones con el fin de ayudar a regenerar el cartílago en áreas como las articulaciones.
Anthony Atala, co-autor junto a Yoo del estudio, comenta en Materia:
Hay sistemas 3D que imprimen células y geles. Pero la impresora 3D que nuestro equipo ha desarrollado es única al poder utilizar tanto geles de biomateriales como polímeros rígidos, pudiendo crear cualquier forma tridimensional.
No sólo eso, según Atala, el futuro se muestra aún más esperanzador:
Podemos imprimir proteínas, factores de crecimiento y otros líquidos en la estructura para ayudar a promover la regeneración una vez implantado. Aunque el dispositivo todavía es experimental, estamos explorando sus posibilidades en órganos como el riñón y tejidos estructurados como el de la oreja.
La clave del éxito de esta impresora modificada es la máquina de electrospinning (o máquina de electrohilado). Ésta es la encargada de crear estructuras porosas gracias a un material sintético, polímeros, a través de una corriente eléctrica. Por tanto, la máquina puede generar fibras muy finas de una solución de polímero, lo que consigue que las células del cartílago real se integren en el tejido circundante.
Ampliar en: ALT1040
Bajo licencia Creative Commons
Smartphones, ideales para propagar las bacterias

Según Jeffrey Cain, presidente de la Academia Americana de Médicos de Familia y jefe de medicina familiar en el Hospital de Niños de Colorado:
“Algunas cosas que creemos que son personales, realmente, son más públicas de lo que pensamos. Las bacterias de un smartphone pueden causar gripe, conjuntivitis o diarrea”
Y, lo peor de esto, es que hay un abismo entre las sustancias limpiadoras que recomiendan los médicos y las que sugieren los fabricantes, ya que estos últimos prohíben casi cualquier sustancia de limpieza cotidiana por peligro de ruptura para nuestras preciadas pantallas móviles, o directamente recomiendan sustancias que no eliminan las bacterias al 100 % (hay enfermedades que con tan solo 10 bacterias provocan una enfermedad…).
Sí, diréis lo que yo he comentado al principio, que “todo tiene bacterias”, como un bolígrafo o un ordenador. Pero, el gran problema es que un smartphones son móviles, si, los transportamos a cualquier sitio: cocina, comedor, reunión, bar o restaurante, ¡incluso algunos lo llevan al gimnasio! Por todo esto, el profesor Michael Schmidt, vicepresidente de microbiología e inmunología de la Universidad Médica de Carolina del Sur afirma:
“Estamos alimentando a los animalitos. Todos hemos visto alguna vez una mancha grasienta en la pantalla táctil del móvil, y donde hay grasa hay bichos”
Para muestra, como siempre un estudio: un laboratorio probó ocho móviles seleccionados al azar de una oficina de Chicago. No encontraron bacterias E. coli o estafilococos, que podría haber sido común, sino que encontraron coliformes, y en números elevados, un tipo de bacteria típico de contaminación fecal (si, es tan malo como suena). Como comparación, en el agua potable el límite de estas bacterias es menos de 1 unidad por cada 100 ml, y en estos móviles había entre 2700 y 4200 unidades de ellas.
Según el Dr. Donald Hendrickson, presidente de los laboratorios HML y profesor de microbiología médica en la Ball State University, los resultados son bastante malos, y demuestran una falta evidente de higiene y de lavado de manos. Probaron también diferentes métodos de limpieza (agua, alcohol, limpiador de cristales y toallitas de limpieza electrónica). Como cabía esperar, el alcohol fue el mejor método, y el agua el peor.
Fuente: Medciencia
Los “virus informáticos” del ADN

Gran parte del ADN está compuesto de transposones, trozos de ADN que actúan como “virus informáticos” capaces de moverse y de copiarse a sí mismos. Gran parte de la “actividad bioquímica específica” observada por el proyecto ENCODE podría ser resultado de la actividad de estos “virus informáticos” que plagan nuestro genoma como el “spam” lo hace en nuestro correo electrónico. Te recomiendo leer Sean Eddy, ”ENCODE says what?,” Cryptogenomicon, September 8th, 2012. Sobre los errores que yo mismo en este blog y gran parte de los medios hemos cometido a la hora de interpretar los resultados de ENCODE también recomiendo Mike White, “ENCODE Media FAIL (or, Where’s the Null Hypothesis?),” The Finch & Pea’s, 6 September 2012.
En casa tienes dos tipos de “basura” bien diferenciadas; por un lado, ese revoltijo de objetos que una vez te fueron útiles, pero ahora no lo son, que guardas porque te traen recuerdos o simplemente por que sí; y por otro lado, las cosas que deseas desechar, que consideras desperdicios que dentro de unas horas acabarán en la bolsa de basura y en el contenedor de basura de la esquina. En ciertas ocasiones ambos tipos de “basura” se encuentran a tu alrededor, incluso pueden llegar a confundirse.
El tamaño no siempre importa, al menos respecto al ADN. Dos especies similares evolutivamente pueden tener genomas de tamaño muy diferente. La diferencia está en la cantidad de transposiciones que contienen. Los transposones, como ilustra el vídeo, son elementos o trozos de ADN que actúan como “virus informáticos” moviéndose por el resto del ADN, copiándose a sí mismos, dentro del genoma del huésped. Barbara McClintock recibió el Nobel de Fisiología o Medicina en 1983 por su descubrimiento.
Sean Eddy nos recuerda que casi el 10% del genoma humano está compuesto por casi un millón de copias del transposón Alu, que contiene unas 300 bases. Estos transposones están relacionados con ciertos virus y se cree que son parásitos del ADN. Se infiltran en el genoma, se reproducen, se multiplican, se difunden, y acaban muriendo, mutando o decayando, dejando como rastro del pasado ciertas secuencias de bases. En general, los Alu saltan de un lugar a otro dentro de la parte no codificante del ADN que podemos llamar “ADN basura” y, que se sepa, no tiene ningún efecto apreciable sobre nosotros. Algunos animales, como las salamandras, tiene su ADN repleto de transposones, por ello su longitud es unas diez mayor que la nuestra.
Lo que hay que tener claro es que solo el 1% del ADN codifica proteínas (son genes en sentido estricto), que entre el 1-4% no codificante actúa como regulador de la expresión de los genes, y que entre el 40-50% son transposones a los que podríamos llamar “ADN basura.” El ADN restante, entre el 40-50% todavía no se sabe muy bien qué papel juega o si tienen alguna “función” específica. El objetivo del proyecto ENCODE es aclararlo, pero los resultados publicados hasta ahora son un primer paso y no se puede afirmar que el término “función bioquímica” implica un cambio en el fenotipo o una “función biológica” en sentido estricto.
Hay una gran diferencia entre los dos tipos de “basura” que tienes en tu casa, la que acabará como desperdicio y la que atesorarás por mucho tiempo. Esta última puede que un día acabe como desperdicio, o puede que le encuentres alguna utilidad práctica. Gran parte del “ADN basura” puede ser de este segundo tipo y quizás la evolución acabe dándole una “función biológica” algún día.
Artículo completo en: Francis (th)E mule Science’s News
¿Será el ADN el soporte de almacenamiento definitivo?
La cantidad de información que produce la humanidad sigue creciendo y su preservación para las generaciones futuras se vuelve problemática. Una posible solución implica el almacenamiento en el ADN. Un grupo de investigadores norteamericanos ilustra el potencial del método mediante el registro de un libro entero en sólo un picogramo de ADN.
Nuestro mundo se está volviendo más y más información a través los datos que circulan a través de los ordenadores e internet, consecuencias de la obra de Alan Turing . Fotos, vídeos, textos, datos digitales de todo tipo, su cantidad se duplica cada año debido a la actividad del Homo sapiens . Pero, ¿cuánta de esta información estará disponible para la próxima generación y cómo almacenarla de forma duradera y discreta? Debido a que la humanidad ha producido en 2011 unos 10 21 bytes de información, y esta cifra se habrá multiplicado por 50 en el año 2020. ¿Cómo almacenar los registros médicos, la música u otras obras de arte con materiales que pueden durar por lo menos un siglo, por ejemplo?
El trabajo sobre este tema de almacenamiento de archivos de la humanidad ha avanzado en los últimos años como lo demuestra, por ejemplo, el M-Disc . Pero una de las técnicas más prometedoras parece que es basándose en el ADN (DNA). Esta idea se explora desde hace algún tiempo y un artículo reciente en Science ilustra el poder de almacenar información digital en la mítica molécula de la vida, cuya estructura fue elucidada por Watson y Crick hace casi 60 años.
Uno de los autores del artículo de Science no es otro que George Church , bien conocido por su trabajo en biología sintética . Este es su libro, Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves, que fue grabado y leído con una nueva técnica que consiste en un soporte de hebras de ADN. El libro en sí contiene 53426 palabras, 11 imágenes y un programa JavaScript que constituyen una riqueza de información de 5.37 Mbit. Una millonésima de la millonésima parte de un gramo de ADN fue suficiente para asegurar su almacenamiento. El récord anterior de ADN fue 7920 bit. Tiene casi 1000 veces de cantidad de información almacenada.
Millones de gigabit por centímetro cúbico de ADN
Este volumen de información no tiene nada de extraordinario en sí mismo. Pero la densidad de almacenamiento es espectacular, ya que es equivalente a 5.5 millones petabit o un gigabit por centímetro cúbico. Esta es mucho mayor que la de los discos duros y más de 10 millones de veces la densidad de almacenamiento de un CD . Sin embargo, el almacenamiento de ADN obtenido por los investigadores no pueden competir con el disco duro porque no se puede leer, escribir o borrar la información a voluntad.
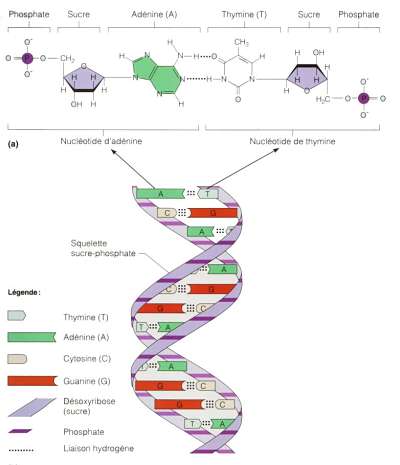
Para almacenar la información, es necesario sintetizar cadenas de ADN en la que se almacenan los datos en forma binaria en nucleótidos de adenina (A), timina (T), citosina (C) y guanina (G). Cada hebra de ADN es un fragmento de la información total almacenada en un soporte de vidrio . Un código así contenido en la secuencia de nucleótidos indica a qué parte del archivo, por ejemplo que contiene el libro de George Churh, en la cadena de ADN. Finalmente, se debe utilizar la técnica de secuenciación de ADN y tratar la información obtenida del proceso en la computadora para recuperar la información original. Procesar poco práctico y caro, obviamente. Esta es la razón por la que el almacenamiento con ADN está más bien destinado a archivar datos. No parecen destinadas a reemplazar las memorias de nuestros ordenadores en la vida cotidiana.
La técnica no implica usar el ADN de las células vivas (se correría el riesgo de alterar la información registrada, por mutación), y como el ADN del pasado puede mantenerse intacto durante miles de años a temperatura ambiente, parece probable que los archivos del futuro de la humanidad, en efecto constarán de ADN. Esto es sorprendente si se considera que lo mismo es cierto para la información genética de las especies vivas.
Fuente: Futura-Sciences
Fusión de lo biológico y lo electrónico

Investigadores desarrollan método para hacer crecer tejidos ‘Cyborg’ con nanoelectrónica embebida.
Científicos de Harvard, por primera vez, han creado un tipo de tejido «cyborg» mediante la incorporación de una red tridimensional funcional, de cables biocompatibles a nanoescala en tejidos humanos «ingenierizados».
Como se describe en un artículo publicado el 26 de agosto en Nature Materials, un equipo de investigación multiinstitucional liderado por Charles M. Lieber, el Hyman Mark, Jr. Profesor de Química en la Universidad de Harvard y Daniel Kohane, profesor de Harvard Medical School en el Departamento de Anestesia en el Hospital infantil de Boston desarrollaron un sistema para la creación de «andamios» a nanoescala que podrían ser sembrados con células que más tarde se convirtieran en tejido.
También contribuyeron a la obra Robert Langer, del Instituto de Koch en el Massachusetts Institute of Technology, y Suo Zhigang, E. Allen, y Marilyn M. Puckett profesor de Mechanics and Materials at Harvard’s School of Engineering and Applied Sciences.
«Los métodos actuales que tenemos para el seguimiento o la interacción con los sistemas vivos son limitados», dijo Lieber. «Podemos usar electrodos para medir la actividad en las células o tejidos, pero las daña. Con esta tecnología, por primera vez, podemos trabajar en la misma escala que la unidad de sistema biológico sin interrumpirlo. En última instancia, se trata de la fusión de tejido con la electrónica de una manera que se hace difícil determinar dónde termina el tejido y empieza la electrónica. »
La investigación responde a la preocupación que ha sido asociada con el trabajo sobre el tejido de bioingeniería – cómo crear sistemas capaces de detectar los cambios químicos o eléctricos en el tejido después de haber sido cultivados e implantados. El sistema también podría presentar una solución a las luchas de los investigadores en el desarrollo de métodos para estimular directamente los tejidos artificiales y medir las reacciones celulares.
«En el cuerpo, el sistema nervioso autónomo realiza un seguimiento de pH, química, oxígeno y otros factores, y desencadena respuestas según sea necesario,» explicó Kohane. «Necesitamos ser capaces de imitar el tipo de bucles de retroalimentación intrínseca que el cuerpo ha desarrollado con el fin de mantener el control de calidad en el nivel celular y tisular.»
Utilizando el sistema nervioso autónomo como inspiración, Bozhi Tian, un antiguo estudiante de doctorado bajo Lieber y exestudiante postdoctoral en el Kohane y los laboratorios de Langer, y colaborador Jia Liu trabajaron en el laboratorio de Lieber en Harvard para construir una malla como las redes de silicio a nanoescal,a cables-sobre 30 a 80 nm de diámetro, en forma de planos lisos o en una conformación reticular. El proceso de construcción de las redes, Lieber, dijo, es similar a la utilizada para grabar microchips.
Comenzando con un sustrato de dos dimensiones, los investigadores diseñaron una malla de polímero orgánico alrededor de los cables a nanoescala, que sirven como los elementos críticos de detección a nanoescala. Electrodos a nanoescala, que conectan los elementos de nanocables, se construyeron entonces dentro de la malla para permitir los transistores de nanocables para medir la actividad en las células sin dañarlas. Una vez completo, el sustrato se disolvió, dejando a los investigadores con una esponja en forma de red o una malla que puede ser doblado o enrollado en un huésped de formas tridimensionales.
Una vez completa, las redes eran lo suficientemente porosas para permitir el equipo de semillas con células y estimular las células para crecer en cultivos 3D.
«Los esfuerzos previos para crear redes de bioingeniería de sensores se han centrado en dos dimensiones, donde los diseños de células de cultivo crecen en la parte superior de los componentes electrónicos, o en los diseños conformados donde las sondas se colocan en superficies de tejido», dijo Tian. «Es deseable tener una imagen precisa del comportamiento celular dentro de la estructura 3D de un tejido, y también es importante tener sondas a nanoescala para evitar la interrupción de cualquiera arquitectura celular o tisular.»
Usando las células del corazón y de los nervios, el equipo ha dirigido con éxito que los tejidos contengan incrustadas redes a nanoescala sin afectar la viabilidad de las células o la actividad. Los dispositivos integrados, que fueron capaces de detectar señales eléctricas generadas por las células profundas dentro del tejido, y para medir los cambios de estas señales en respuesta a los fármacos cardio-o neuro-estimulantes.
Los investigadores también fueron capaces de construir vasos sanguíneos de bioingeniería, y se utiliza la tecnología integrada para medir los cambios de pH – como se vería en respuesta a la inflamación, isquemia y otros entornos de bioquímica o celular – tanto dentro como fuera de los vasos.
A pesar de una serie de aplicaciones potenciales existentes para esta tecnología, el uso a más corto plazo, dijo Lieber, puede provenir de la industria farmacéutica, donde los investigadores pueden utilizar la tecnología para estudiar con más precisión cómo las drogas recién desarrollados actuan en tejidos tridimensionales, en lugar de capas delgadas de células cultivadas. El sistema también podría usarse algún día para controlar los cambios en el cuerpo y reaccionar en consecuencia, sea a través de la estimulación eléctrica o la liberación de un fármaco.
Fuente: EurekAlert!
Libro escrito en ADN
Un transistor tenía el tamaño de una aspirina a mediados del siglo XX, pero mide lo que un virus a principios del XXI. Esta increíble historia del transistor menguante, uno de los grandes logros de la tecnología moderna, es lo que subyace a la llamada ley de Moore: que la potencia de los chips se duplica cada año y medio. La palabra ley en este caso no se usa en el sentido de un principio científico universal e inviolable, como la primera ley de Newton o la segunda ley de la termodinámica, sino más bien como en la ley de Murphy, en un sentido jocoso y provisional.
Los inventores del almacenamiento de información no fuimos los humanos. Fue la naturaleza. El ADN, la famosa doble hélice que constituye el soporte de la información genética, es precisamente la base de datos que utilizamos todos los seres vivos desde hace 3000 millones de años.
Dos genetistas de la Universidad de Harvard acaban de dejar al mundo boquiabierto al escribir un libro en la molécula de ADN. En vez de una ristra de ceros y unos, el resultado es una secuencia de a, t, g y c, las bases o letras con que se escriben los genes, pero la idea es básicamente la misma. Y el ADN es el soporte de información más compacto que conoce la tecnología contemporánea, con una densidad de almacenamiento un millón de veces superior a los actuales discos duros.
Fuente: ElPaís.com

autobus las palmas aeropuerto cetona de frambuesa