Actualidad informática
Noticias y novedades sobre informática
Ordenadores orgánicos de ADN podrían procesar datos en nuestros cuerpos
Siempre imaginamos dispositivos electrónicos que se fabrican a partir de chips de silicio, con la que los ordenadores almacenan y procesar información en forma de dígitos binarios (ceros y unos) representados por pequeñas cargas eléctricas. Pero no necesariamente tiene que ser de esta manera: Entre las alternativas al silicio están los medios orgánicos tales como el ADN.
La computación de ADN se demostró por primera vez en 1994 por Leonard Adleman, que codificó y se resolvió el problema del viajante, un problema de matemáticas para encontrar la ruta más eficiente para un vendedor, entre ciudades, en su totalidad en el ADN.
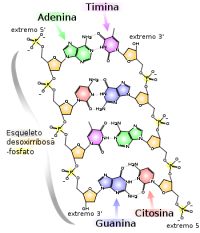
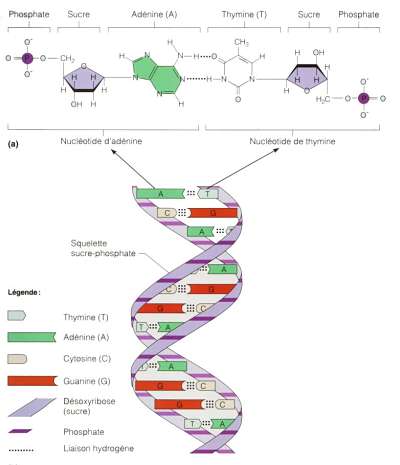
EL ácido desoxirribonucleico, ADN, puede almacenar grandes cantidades de información codificada como secuencias de las moléculas, conocidos como nucleótidos, citosina (C), guanina (G), adenina (A), o timina (T). La complejidad y la enorme variación de los códigos genéticos de diferentes especies demuestra cuánta información puede ser almacenada en el ADN, que se codifica mediante CGAT, y esta capacidad puede ser objeto de uso por parte de la informática. Las moléculas de ADN se pueden emplear para procesar la información, utilizando un proceso de unión entre los pares de ADN conocido como hibridación. Esto lleva a cadenas simples de ADN como entrada y produce hebras de ADN a través de transformación como salida.
Desde el experimento de Adleman, muchas «circuitos» basados en ADN se han propuesto para implementar métodos computacionales, tales como la lógica de Boole, fórmulas aritméticas y cálculo de redes neuronales. Llamada programación molecular, este enfoque aplica conceptos y diseños habituales de la computación a escala nanométrica, siendo apropiado para trabajar con el ADN.
En esta «programación» lo que tiene sentido es realmente la bioquímica. Los «programas» creados son en base a la selección de moléculas que interactúan de una manera que logran un resultado específico en el proceso de autoensamblaje de ADN, donde colecciones desordenadas de moléculas espontáneamente interactúan para formar la disposición deseada de filamentos de ADN.
‘Robots’ de ADN
El ADN también se puede utilizar para controlar el movimiento, lo que permite dispositivos basados en la nanomecánica que usan el ADN. Esto se logró por primera vez por Bernard Yurke y sus colegas en 2000, que crearon a partir de hebras de ADN un par de pinzas que se abrían y pellizcaban. Experimentos posteriores, como el de Shelley Wickham y colegas en 2011 y en el laboratorio de Andrew Turberfield en Oxford demostraron que máquinas para caminar nanomoleculares hechas enteramente de ADN, podrían recorrer rutas establecidas.
Una posible aplicación es que un nanorobot caminante de ADN podría progresar a lo largo de pistas de toma de decisiones y dar la señal cuando se alcanza el final de la pista, lo que indica que el cómputo ha terminado. Al igual que los circuitos electrónicos se imprimen en tarjetas de circuitos, las moléculas de ADN se podrían utilizar para imprimir pistas similares dispuestas en árboles de decisión lógicos en un chip de ADN, y las enzimas se utilizarían para controlar la decisión de ramificación a lo largo del árbol, haciendo que el caminante tome una pista u otra.
Los caminantes de ADN también pueden transportar carga molecular, y así podrían ser utilizados para administrar medicamentos dentro del cuerpo.
¿Por qué la computación de ADN?
Entre las muchas características atractivas de las moléculas de ADN, se incluye su tamaño (ancho 2 nm), programabilidad y alta capacidad de almacenamiento – mucho mayor que sus homólogos de silicio. ADN también es versátil, barato y fácil de sintetizar, y la computación con ADN requiere mucha menos energía que los procesadores de silicio eléctricos.
Su desventaja es la velocidad: en la actualidad lleva varias horas para calcular la raíz cuadrada de un número de cuatro dígitos, algo que una computadora tradicional podría calcular en una centésima de segundo. Otro inconveniente es que los circuitos de ADN son de un solo uso, y necesitan ser recreado para ejecutar el mismo cálculo de nuevo.
Quizás la ventaja más grande de ADN a través de circuitos electrónicos es que puede interactuar con su entorno bioquímico. La computación con moléculas implica reconocer la presencia o ausencia de ciertas moléculas, y por lo que una aplicación natural de la informática de ADN es llevar esa capacidad de programación al ámbito de biosensores ambientales o de la entrega de medicamentos y terapias dentro de los organismos vivos.
Programas de ADN ya se han empleado en usos médicos, tales como el diagnóstico de la tuberculosis. Otro uso propuesto es un «programa» nano-biológica por Ehud Shapiro, del Instituto de Ciencia Weizmann en Israel, denominado el «médico de la célula» que se dirige a moléculas cancerígenas. Otros programas de ADN para aplicaciones médicas linfocitos diana (un tipo de glóbulo blanco), que se definen por la presencia o ausencia de ciertos marcadores de células y así se pueden detectar de forma natural con la lógica booleana verdadera/falso. Sin embargo, se requiere más esfuerzo antes de que podamos inyectar drogas inteligentes directamente en los organismos vivos.
El futuro de la computación de ADN
Tomado en términos generales, el cómputo de ADN tiene un enorme potencial de futuro. Su gran capacidad de almacenamiento, bajo coste energético, facilidad de fabricación que explota el poder de autoensamblaje y su fácil afinidad con el mundo natural es una entrada a la informática a escala nanométrica, posiblemente a través de diseños que incorporan ambos componentes moleculares y electrónicas. Desde su creación, la tecnología ha avanzado a gran velocidad, la entrega de diagnósticos en el punto de atención y prueba de concepto de medicamentos inteligentes – aquellos que pueden tomar decisiones de diagnóstico sobre el tipo de terapia para entregar.
Hay muchos desafíos, por supuesto, que hay que abordar de manera que la tecnología puede avanzar desde el concepto de prueba de drogas inteligentes: la fiabilidad de los caminantes de ADN, la solidez del autoensamblaje de ADN, y la mejora de la entrega de fármacos. Pero tras un siglo de investigación la informática tradicional está bien situada para contribuir al desarrollo de la informática de ADN a través de nuevos lenguajes de programación, abstracciones, y técnicas de verificación formal – técnicas que ya han revolucionado el diseño de circuitos de silicio, y puede ayudar a poner en marcha la computación orgánica por el mismo camino.
Fuente: The Conversation
¿Cuánto ADN hay en la Tierra y cuánta información contiene?
 ¿Cuánto ADN hace falta, entonces, para formar un ser humano? ¿Cuánta información procesa cada ser vivo? Es más, ¿Cuánto ADN en total habrá en el mundo en el que vivimos? Tanto en capacidad de guardar información como en cantidad, las cifras son sencillamente impresionantes.
¿Cuánto ADN hace falta, entonces, para formar un ser humano? ¿Cuánta información procesa cada ser vivo? Es más, ¿Cuánto ADN en total habrá en el mundo en el que vivimos? Tanto en capacidad de guardar información como en cantidad, las cifras son sencillamente impresionantes.
Supongamos que la tierra es un gran sistema de computación. Esto precisamente es lo que han hecho unos investigadores del centro de Astrobiología y la Universidad de Edimburgo. Todo con la intención de medir cuánto ADN podría haber sobre la faz de la Tierra. Según sus estimaciones, en masa, el ADN existente en nuestro pequeño planeta es de unas 5 por 10 elevado a diez (5×10^10) toneladas de ADN. Es decir, unos 1000 millones de enormes contenedores de carga. Lo que parece muchísimo para algo que mide tan poco que no podemos ver a simple vista. Pero si esta cifra no te impresiona, tal vez lo haga su implicación. Puesto que el ADN es la unidad fundamental de información y su misión es almacenarla procesarla, esto 1000 millones de contenedores suponen una capacidad de unas 5.3 × 10^31 (±3.6 × 10^31) megabases (Mb, que equivalen a un millón de pares de bases).
La cantidad de información almacenada excede cualquier posibilidad de imaginarla. Pero para que nos hagamos una idea, el ADN de la Tierra es unas 10 elevado a las 22 (10^22) veces más rápido procesando que el superordenador más rápido de la Tierra, el Tianhe-2 chino, que cuenta con unos 33.86 PetaFLOPS. Esto supone, en concreto, un poder de computación de unos 10 elevado a 15 (10^15) yottaNOPS. Para quien no lo sepa, yotta significa a su vez 10 elevado a 24 (10^24). Además, necesitaríamos unos 10 elevados a la 21 (10^21) ordenadores como éste para almacenar toda la información que guarda el ADN en total. Aunque hasta ahora se había investigado muchísimo sobre la cantidad de ADN total existente en organismos, es la primera vez que alguien intenta estimar la capacidad en información relacionada con cuánto ADN existe.
Ampliar en: Conocer Ciencia
Craig Venter próximo a crear vida sintética

Por primera vez estamos cerca de crear vida artificial desde el principio. Así lo afirma Craig Venter, fundador del J. Craig Venter Institute en Rockville, Maryland (EE.UU.), y famoso por crear la primera célula con un genoma sintético.
«Creemos que estamos cerca, pero no hemos presentado un documento todavía», dijo en la cumbre mundial de los grandes retos en Londres esta semana.
Venter anunció en 2010 que había traído a la vida de una versión casi completamente sintética de la bacteria Mycoplasma mycoides los, al trasplantarla en la cáscara vacía de otra bacteria.La última creación de Venter, que él ha llamado el Ave María Genoma, se hará a partir de cero con los genes que él y sus compañeros de instituto, Clyde Hutchison y Hamilton Smith, consideren indispensable para la vida.
El equipo está usando simulaciones por ordenador para entender mejor lo que se necesita para crear un simple, celular auto-replicante. «Una vez que tengamos un mínimo de chasis podemos añadir algo más», dice.
La búsqueda de Venter para diseñar algas que produzcan más aceite de lo normal también va bien. «Hemos sido capaces de aumentar la fotosíntesis tres veces, lo que significa que tenemos tres veces más energía por fotón [del sol] a partir de algas naturales», afirmó. También anunció que su programa para recorrer los océanos para buscar vida microscópica nueva hasta ahora ha encontrado 80 millones de nuevos genes para la biología.
Fuente: NewScientist
Foto: dfarber via photopin cc
Cuatro gramos de ADN bastarían para almacenar la información del planeta

Investigadores han logrado crear un proceso con el que sería posible almacenar sin errores datos en la forma de cadenas sintéticas de ADN, las cuales en teoría podrían perdurar intactas por miles de años. De acuerdo con sus cálculos, el método de codificación de información desarrollado marcaría la posibilidad de guardar cerca de cien millones de horas de video en Alta Definición, o incluso contener toda la información del mundo, equivalente a 1.8 zettabytes, en tan sólo cuatro gramos de peso.
Hace unos días se mostraban los grandes avances que marcaban este proceso como una realidad prácticamente inevitable y ahora aquí presentamos la primera prueba. El estudio, publicado en la revista de investigación Nature esta misma semana, fue desarrollado por un grupo de científicos británicos del European Bioinformatics Institute EMBL-EBI, quienes indican haber podido almacenar una versión MP3 del famoso discurso de Martin Luther King “I Have a Dream” junto a una imagen del logotipo de la institución y algunos archivos de texto (probablemente listas del súper), dentro de una cadena sencilla de ADN.
Fuente: AlT1040
Almacenamiento de prosa y recuperación libre de errores en ADN
Es uno de los discursos más emblemáticos de todos los tiempos, y ahora ha sido inmortalizado de una forma muy inusual. Un fragmento de 1963 de Martin Luther King «I have a dream» (Tengo un sueño) ha sido almacenado en el alfabeto del ADN.
Nick Goldman en el European Bioinformatics Institute en Hinxton, Reino Unido, y sus colegas sintetizaron ADN para codificar una mezcla ecléctica de información en sus componentes adenina, timina, citosina y guanina. Utilizaron estas «letras» para grabar un archivo de audio de 26 segundos del discurso de King, los 154 sonetos de Shakespeare, una foto digital de su laboratorio y el famoso artículo en el que James Watson y Francis Crick describieron por primera vez la estructura de doble hélice del ADN .
El equipo construyó anteriormente técnicas de codificación de ADN añadiendo corrección de errores, permitiendo la recuperación con el 100 por cien de precisión.
Memoria basada en el ADN es eficaz dado que el ADN puede durar miles de años sin condiciones especiales de almacenamiento, aparte de encontrarse en algún lugar frío, oscuro y seco. En teoría, el ADN puede codificar o menos la capacidad de 100 000 millones de DVD por gramo de ADN de cadena sencilla, por lo que es potencialmente útil para almacenar la enorme cantidad de datos archivados producidos por lugares como el CERN.
Los “virus informáticos” del ADN

Gran parte del ADN está compuesto de transposones, trozos de ADN que actúan como “virus informáticos” capaces de moverse y de copiarse a sí mismos. Gran parte de la “actividad bioquímica específica” observada por el proyecto ENCODE podría ser resultado de la actividad de estos “virus informáticos” que plagan nuestro genoma como el “spam” lo hace en nuestro correo electrónico. Te recomiendo leer Sean Eddy, ”ENCODE says what?,” Cryptogenomicon, September 8th, 2012. Sobre los errores que yo mismo en este blog y gran parte de los medios hemos cometido a la hora de interpretar los resultados de ENCODE también recomiendo Mike White, “ENCODE Media FAIL (or, Where’s the Null Hypothesis?),” The Finch & Pea’s, 6 September 2012.
En casa tienes dos tipos de “basura” bien diferenciadas; por un lado, ese revoltijo de objetos que una vez te fueron útiles, pero ahora no lo son, que guardas porque te traen recuerdos o simplemente por que sí; y por otro lado, las cosas que deseas desechar, que consideras desperdicios que dentro de unas horas acabarán en la bolsa de basura y en el contenedor de basura de la esquina. En ciertas ocasiones ambos tipos de “basura” se encuentran a tu alrededor, incluso pueden llegar a confundirse.
El tamaño no siempre importa, al menos respecto al ADN. Dos especies similares evolutivamente pueden tener genomas de tamaño muy diferente. La diferencia está en la cantidad de transposiciones que contienen. Los transposones, como ilustra el vídeo, son elementos o trozos de ADN que actúan como “virus informáticos” moviéndose por el resto del ADN, copiándose a sí mismos, dentro del genoma del huésped. Barbara McClintock recibió el Nobel de Fisiología o Medicina en 1983 por su descubrimiento.
Sean Eddy nos recuerda que casi el 10% del genoma humano está compuesto por casi un millón de copias del transposón Alu, que contiene unas 300 bases. Estos transposones están relacionados con ciertos virus y se cree que son parásitos del ADN. Se infiltran en el genoma, se reproducen, se multiplican, se difunden, y acaban muriendo, mutando o decayando, dejando como rastro del pasado ciertas secuencias de bases. En general, los Alu saltan de un lugar a otro dentro de la parte no codificante del ADN que podemos llamar “ADN basura” y, que se sepa, no tiene ningún efecto apreciable sobre nosotros. Algunos animales, como las salamandras, tiene su ADN repleto de transposones, por ello su longitud es unas diez mayor que la nuestra.
Lo que hay que tener claro es que solo el 1% del ADN codifica proteínas (son genes en sentido estricto), que entre el 1-4% no codificante actúa como regulador de la expresión de los genes, y que entre el 40-50% son transposones a los que podríamos llamar “ADN basura.” El ADN restante, entre el 40-50% todavía no se sabe muy bien qué papel juega o si tienen alguna “función” específica. El objetivo del proyecto ENCODE es aclararlo, pero los resultados publicados hasta ahora son un primer paso y no se puede afirmar que el término “función bioquímica” implica un cambio en el fenotipo o una “función biológica” en sentido estricto.
Hay una gran diferencia entre los dos tipos de “basura” que tienes en tu casa, la que acabará como desperdicio y la que atesorarás por mucho tiempo. Esta última puede que un día acabe como desperdicio, o puede que le encuentres alguna utilidad práctica. Gran parte del “ADN basura” puede ser de este segundo tipo y quizás la evolución acabe dándole una “función biológica” algún día.
Artículo completo en: Francis (th)E mule Science’s News
¿Será el ADN el soporte de almacenamiento definitivo?
La cantidad de información que produce la humanidad sigue creciendo y su preservación para las generaciones futuras se vuelve problemática. Una posible solución implica el almacenamiento en el ADN. Un grupo de investigadores norteamericanos ilustra el potencial del método mediante el registro de un libro entero en sólo un picogramo de ADN.
Nuestro mundo se está volviendo más y más información a través los datos que circulan a través de los ordenadores e internet, consecuencias de la obra de Alan Turing . Fotos, vídeos, textos, datos digitales de todo tipo, su cantidad se duplica cada año debido a la actividad del Homo sapiens . Pero, ¿cuánta de esta información estará disponible para la próxima generación y cómo almacenarla de forma duradera y discreta? Debido a que la humanidad ha producido en 2011 unos 10 21 bytes de información, y esta cifra se habrá multiplicado por 50 en el año 2020. ¿Cómo almacenar los registros médicos, la música u otras obras de arte con materiales que pueden durar por lo menos un siglo, por ejemplo?
El trabajo sobre este tema de almacenamiento de archivos de la humanidad ha avanzado en los últimos años como lo demuestra, por ejemplo, el M-Disc . Pero una de las técnicas más prometedoras parece que es basándose en el ADN (DNA). Esta idea se explora desde hace algún tiempo y un artículo reciente en Science ilustra el poder de almacenar información digital en la mítica molécula de la vida, cuya estructura fue elucidada por Watson y Crick hace casi 60 años.
Uno de los autores del artículo de Science no es otro que George Church , bien conocido por su trabajo en biología sintética . Este es su libro, Regenesis: How Synthetic Biology Will Reinvent Nature and Ourselves, que fue grabado y leído con una nueva técnica que consiste en un soporte de hebras de ADN. El libro en sí contiene 53426 palabras, 11 imágenes y un programa JavaScript que constituyen una riqueza de información de 5.37 Mbit. Una millonésima de la millonésima parte de un gramo de ADN fue suficiente para asegurar su almacenamiento. El récord anterior de ADN fue 7920 bit. Tiene casi 1000 veces de cantidad de información almacenada.
Millones de gigabit por centímetro cúbico de ADN
Este volumen de información no tiene nada de extraordinario en sí mismo. Pero la densidad de almacenamiento es espectacular, ya que es equivalente a 5.5 millones petabit o un gigabit por centímetro cúbico. Esta es mucho mayor que la de los discos duros y más de 10 millones de veces la densidad de almacenamiento de un CD . Sin embargo, el almacenamiento de ADN obtenido por los investigadores no pueden competir con el disco duro porque no se puede leer, escribir o borrar la información a voluntad.
Para almacenar la información, es necesario sintetizar cadenas de ADN en la que se almacenan los datos en forma binaria en nucleótidos de adenina (A), timina (T), citosina (C) y guanina (G). Cada hebra de ADN es un fragmento de la información total almacenada en un soporte de vidrio . Un código así contenido en la secuencia de nucleótidos indica a qué parte del archivo, por ejemplo que contiene el libro de George Churh, en la cadena de ADN. Finalmente, se debe utilizar la técnica de secuenciación de ADN y tratar la información obtenida del proceso en la computadora para recuperar la información original. Procesar poco práctico y caro, obviamente. Esta es la razón por la que el almacenamiento con ADN está más bien destinado a archivar datos. No parecen destinadas a reemplazar las memorias de nuestros ordenadores en la vida cotidiana.
La técnica no implica usar el ADN de las células vivas (se correría el riesgo de alterar la información registrada, por mutación), y como el ADN del pasado puede mantenerse intacto durante miles de años a temperatura ambiente, parece probable que los archivos del futuro de la humanidad, en efecto constarán de ADN. Esto es sorprendente si se considera que lo mismo es cierto para la información genética de las especies vivas.
Fuente: Futura-Sciences
Libro escrito en ADN
Un transistor tenía el tamaño de una aspirina a mediados del siglo XX, pero mide lo que un virus a principios del XXI. Esta increíble historia del transistor menguante, uno de los grandes logros de la tecnología moderna, es lo que subyace a la llamada ley de Moore: que la potencia de los chips se duplica cada año y medio. La palabra ley en este caso no se usa en el sentido de un principio científico universal e inviolable, como la primera ley de Newton o la segunda ley de la termodinámica, sino más bien como en la ley de Murphy, en un sentido jocoso y provisional.
Los inventores del almacenamiento de información no fuimos los humanos. Fue la naturaleza. El ADN, la famosa doble hélice que constituye el soporte de la información genética, es precisamente la base de datos que utilizamos todos los seres vivos desde hace 3000 millones de años.
Dos genetistas de la Universidad de Harvard acaban de dejar al mundo boquiabierto al escribir un libro en la molécula de ADN. En vez de una ristra de ceros y unos, el resultado es una secuencia de a, t, g y c, las bases o letras con que se escriben los genes, pero la idea es básicamente la misma. Y el ADN es el soporte de información más compacto que conoce la tecnología contemporánea, con una densidad de almacenamiento un millón de veces superior a los actuales discos duros.
Fuente: ElPaís.com
Por primera vez se simula en ordenador el genoma de un ser vivo
En una movida que promete traer las ventajas de diseño asistido por ordenador (CAD) a los ingenieros genéticos, el primer modelo de computadora de una bacteria completa se ha producido en los EE.UU.. Esto significa que los investigadores pronto será capaces de modificar los modelos de genoma de un organismo en una pantalla de ordenador – o crear formas de vida artificiales – sin los riesgos de llevar a cabo la biología en los laboratorios de biotecnología segura.
El agente patógeno se llama Mycoplasma genitalium, una bacteria implicada en una serie de infecciones uretrales y vaginales. Según los investigadores de la Universidad de Stanford en California, debido a que tiene el genoma más pequeño de cualquier organismo vivo, con sólo 525 genes. Por el contrario, el patógeno popular de laboratorio E. coli tiene 4288 genes.
La modelación se llevó a cabo por el bioingeniero Markus Covert y sus colegas. Para obtener los datos en bruto para su modelo, llevó a cabo una revisión exhaustiva de la literatura – que abarca 900 artículos de investigación – para permitir que el programa modelara 1900 comportamientos observados experimentalmente y las interacciones moleculares que M. genitalium puede tomar parte en durante su ciclo de vida.
En términos de software, se encontraron con los comportamientos de 525 genes podrían ser descritos por 28 algoritmos, cada uno rige el comportamiento de un módulo de software de modelado de un proceso biológico diferente. «Estos módulos se comunican entre sí después de cada paso de tiempo, para hacer un todo unificado que igualan en el mundo real el comportamiento de M. genitalium«, afirma el equipo de Stanford en un comunicado. Su investigación aparece en la revista Cell (doi: 10.1016/j.cell.2012.05.044).
Estos modelos en última instancia, darán a los biólogos la libertad de emprender «¿qué pasaría si» los escenarios más comunes en la ingeniería -el cambio de parámetros en el diseño del genoma, dicen, al igual que un ingeniero civil ajusta el ancho de tablero de un puente en una computadora para ver qué pasa. Además de ser experimentalmente útil, permitiendo organismos artificiales y formas de vida sintéticas que se creen virtualmente (sin daño a nadie), sino que también podría impulsar la seguridad de la biotecnología mediante la prevención de las creaciones accidentales de patógenos letales. En el año 2001, por ejemplo, investigadores en Australia accidentalmente crearon una cepa mortal de la viruela del ratón.
En un artículo de comentario en Cell, los sistemas de los biólogos Peter Freddolino y Saeed Tavazoie de la Universidad de Columbia dicen que esperan que el trabajo pronto se extenderá a los organismos de laboratorio de uso más común como la E. coli -, pero también advierten que la precisión de la técnica aún no se ha demostrado. No está claro, dicen, «qué tan bien los comportamientos generales se puede predecir a partir de una colección de parámetros obtenidos por separado», deducidas a partir de cientos de trabajos de investigación.
Pero el Institutos Nacionales de Salud de EE.UU., que financió el trabajo de modelado, está entusiasmado. Se cree que el modelo es un gran paso hacia la búsqueda de «nuevos enfoques para el diagnóstico y tratamiento de la enfermedad», dice James Anderson, director del programa.
Fuente: NewScientist
Codifican memoria regrabable en el ADN
Investigadores han codificado una forma de memoria regrabable en el ADN. Intentaron crear un sistema de memoria regrabable al dividir elementos genéticos de un bacteriófago -un virus que infecta bacterias- en el ADN de la bacteria Escherichia coli. El artículo fue publicado en Procedings of the National Academy of Sciences y reseñado en Nature.
El uso de sistemas biológicos sintéticos en investigación, asistencia sanitaria, y fabricación, a menudo requiere de la comportamiento histórico autónomo dependiente y por lo tanto, alguna forma de ingeniería de memoria biológica. Por ejemplo, el estudio o la reprogramación de envejecimiento, el cáncer o el desarrollo se beneficiarían de los contadores codificados genéticamente capaces de grabar hasta la división celular de varios cientos o fenómenos de diferenciación. Aunque el material genético en sí mismo constituye un medio natural de almacenamiento de datos, herramientas que permitan a los investigadores de forma fiable y reversible escribir información en el ADN in vivo son escasas. En este sentido, se muestra un módulo datos regrabables direccionables de recombinasa (RAD), que almacena la información digital de forma fiable dentro de un cromosoma. RAD usa módulos de serina de la integrasa y las funciones de excision se adaptan a partir de bacteriófagos para invertir y recuperar secuencias específicas de ADN.
Nuestro elemento de memoria núcleo RAD es capaz de almacenar información pasiva en ausencia de expresión de genes heterólogos por más de 100 divisiones celulares y se puede conmutar repetidamente sin degradación del rendimiento, como se requiere para soportar el almacenamiento de datos. También demuestran cómo la estocasticidad programada en el rendimiento del sistema RAD derivada de la recombinación bidireccional se puede lograr y ajustar por la variación de las tasas de síntesis y degradación de la proteína recombinasa. Las funciones serina recombinasa utilizadas aquí no requieren células específicas de cofactores y deberían ser útiles en la extensión de los métodos de cómputo y control para el estudio e ingeniería de muchos sistemas biológicos.
Ampliar en: Rewritable digital data storage in live cells via engineered control of recombination directionality

autobus las palmas aeropuerto cetona de frambuesa